Datahåndering II: Håndtere og visualisere data#
Læringsutbytte
Etter å ha arbeidet med dette temaet, skal du kunne:
lese data fra fil
rydde og sortere data
visualisere fildata
In God we trust; all others must bring data.
—Statistiker W. Edwards Deming (1900–1993)
Data er over alt. Til daglig prosesserer vi store mengder med data i hjernen vår, og vi lærer og konstruerer nye oppfatninger og sammenhenger basert på disse inntrykkene. Dersom vi snubler i en høy dørterskel tilstrekkelig mange ganger, lærer vi etter hvert å ta høyere skritt over akkurat denne terskelen. Vi klarer også stort sett å kjenne igjen dyr som hunder, katter og aper, og vi klarer å skille dem fra hverandre. Det er fordi vi har samlet masse data i løpet av livet som gjør oss egnet til å trekke slutninger og gjøre (som regel?) gode valg. Samfunnet og internett samler også my data om deg, blant annet for å tilpasse seeropplevelser på Netflix eller annonser på Google. Heldigvis er datainnsamling ganske regulert gjennom lover og regler (GDPR = General Data Protection Regulation).
Vi kan også bruke data til å illustrere sammenhenger i natur og samfunn, og som utgangspunkt for modellering. Vi skal derfor her se på hvordan vi kan lese, bearbeide og visualisere data på en god måte.
Datafiler#
Vi ønsker ofte å oppbevare og overføre data som er i råtekstformat fordi de er robuste og kan leses av alle. Det betyr at dataene ikke har noen formatering eller annen informasjon enn de faktiske dataene. En Word-fil er for eksempel ikke råtekst, fordi den inneholder formatering av tekst som farger, kursivering og tekststørrelse. Eksempler på råtekstfiler er .txt-filer og .csv-filer. Råtekstdata kan vi lage manuelt, eller vi kan få dem fra sensorer eller laste dem ned fra internett. De fleste store datafiler lagres i råtekst.
Vi kan bruke Python til å lese slike data på mange ulike måter. Vi skal se på tre ulike metoder å gjøre det på, slik at du kan benytte det som er best i en gitt situasjon. Alle metodene har fordeler og ulemper, som vi også skal adressere. Vi kommer derimot til å bruke metoden fra det mye brukte Pandas-biblioteket som standard seinere, så det kan være en fordel at du lærer deg denne metoden best. Det er også den enkleste måten å lese filer på.
Vi tar utgangspunkt i en liten fil med få datapunkter, slik at det er enkelt å se hva som skjer når vi leser fila. Fila beskriver temperaturen i en kaffekopp (i \(^oC\)) med tida (i minutter), og ser slik ut:
tid (min), temperatur (grader celsius)
0,90
1,80
2,72
3,64
4,59
5,51
6,45
7,42
8,39
9,37
10,36
Vi ser at fila skiller datapunktene med komma, og at første linje som fungerer som overskrift. Dette er viktig informasjon når vi skal lese fila. For å lese en datafil, må den enten ligge i samme mappe som programmet som leser fila, eller så må du spesifisere hvilken filbane fila har. Det enkleste er å legge den i samme mappe som programmet, eller i en mappe som for eksempel heter “datafiler”, som ligger i samme mappe som programmet ditt.
Nedenfor ser vi tre måter å lese filer på. Vi skal primært bruke Pandas-metoden videre, men gjennomgå alle metodene slik at du kjenner til litt ulike framgangsmåter.
Vi kan bruke grunnstrukturer i Python til å lese filer uten å bruke biblioteker. Da må vi benytte løkker for å gjenta operasjoner for hver linje i fila:
Her bruke vi kommandoen open, med nøkkelordet “r” (read). I løkka leser programmet hver linje, og deler dataene ved hvert komma, spesifisert i kommandoen split. Dette genererer ei liste med to elementer, siden det er to elementer på hver rad. Disse elementene blir tolket som tekst. Derfor konverterer vi dem til flyttall og legger dem i hver sin liste, som vi plotter til slutt. Vi lukker også fila til slutt.
Underveisoppgave
Kjør programmet nedenfor i Trinket-vinduet ovenfor. Hva viser programmet deg?
import matplotlib.pyplot as plt
fil = open("temperatur.txt", 'r')
fil.readline()
t = []
T = []
for rad in fil:
print("rad:", rad)
data = rad.split(",")
print("splittet rad:", data)
print("radelement 1:", data[0], "radelement 2:", data[1])
fil.close()
Det finnes også en nyttig funksjon som heter loadtxt i numpy-biblioteket, som lar deg lese filer på en vektorisert måte uten løkker. Da lages det en array av dataene, med en array for hver kolonne inni denne arrayen. Arrayen er altså todimensjonal, og vi må derfor trekke ut de relevante kolonnene i hver sin endimensjonale array.
Her utfører vi “array-slicing”, det vil si at vi plukker ut elementer fra en todimensjonal array og lager en ny endimensjonal array av det.
Underveisoppgave
Kjør programmet nedenfor i Trinket-vinduet ovenfor. Hva viser programmet deg om array-slicing? Eksperimenter gjerne med å bytte ut verdiene, slik at du forstår hvordan verdiene plukkes ut
import numpy as np
import matplotlib.pyplot as plt
data = np.loadtxt("temperatur.txt", skiprows = 1, delimiter = ",") # Får to arrayer (kolonner) i en array
print(data)
t = data[0:3,0]
T = data[0:3,1]
print(t)
print(T)
plt.scatter(t, T)
plt.xlabel("Tid (s)")
plt.xlabel("Temperatur ($^o$C)")
plt.show()
Et svært mye brukt bibliotek er Pandas-biblioteket. Det benyttes mye i datahåndtering og maskinlæring, og er kanskje den enkleste måten å lese filer på. Med funksjonen read_csv leses filer av typen .txt eller .csv, og vi får en ny datatype som kalles en dataramme (dataframe). En slik datatype kan ses på som en slags dictionary, der kolonneoverskriftene fungerer som nøkler. Dermed skriver vi data[“temperatur”] for å få tilgang til kolonnen med overskriften “temperatur”.
Pandas gir penest og ryddigst output dersom du bruker det i Jupyter Notebook. Vi skal bruke Pandas videre her, så da får du en smakebit på hva som er mulig.
Håndtere data med Pandas#
Noen ganger trenger vi å rydde, utforske og omstrukturere datasettene våre før vi visualiserer dem. Dette egner Pandas seg svært godt til. La oss se på et eksempel. Datafila penguins.txt inneholder ulik informasjon om pingviner som er registrert på ulike øyer. Datafila ligger her i en mappe data som ligger i samme mappe som programmet vårt. Vi leser av fila slik:
import pandas as pd
pingvindata = pd.read_csv("data/penguins.txt", delimiter = ",")
Vi kan ta en kikk på de første eller siste linjene i datasettet vårt ved å bruke funksjonene head eller tail. Dersom vi ikke gir disse funksjonene en parameterverdi, får vi de 5 første eller siste linjene:
pingvindata.head()
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | |
|---|---|---|---|---|---|---|---|
| 0 | Adelie | Torgersen | 39.1 | 18.7 | 181.0 | 3750.0 | MALE |
| 1 | Adelie | Torgersen | 39.5 | 17.4 | 186.0 | 3800.0 | FEMALE |
| 2 | Adelie | Torgersen | 40.3 | 18.0 | 195.0 | 3250.0 | FEMALE |
| 3 | Adelie | Torgersen | NaN | NaN | NaN | NaN | NaN |
| 4 | Adelie | Torgersen | 36.7 | 19.3 | 193.0 | 3450.0 | FEMALE |
Her ser vi både kolonnenavnene og verdier til de fem første pingvinene. Vi kan også se at vi får en litt merkelig verdi, nemlig NaN. Dette står for “Not a Number” og er en vanlig måte å markere at vi mangler data. Det betyr for eksempel at pingvinene er registrert, men at vi ikke fikk undersøkt den. Vi ser for øvrig at read_csv fra Pandas kan lese både NaN-verdier, tekst og tall samtidig.
Rydde data#
Mange ganger har vi behov for å utforske og rydde litt i datasettet vårt. Det kan hende vi kun trenger noen utvalgte verdier, eller det kan hende vi ønsker å slette noe. Programmet nedenfor viser deg noen muligheter. Husk at hvis du skal lagre verdier, må du tilordne dem til variabler. For eksempel finner vi kun damepingvinene ved å skrive pingvindata[pingvindata[“sex”] == “FEMALE”]. Hvis vi vil lage en dataframe som kun inneholder damepingvinene, må vi skrive pingvindata_damer = pingvindata[pingvindata[“sex”] == “FEMALE”].
# Sjekke hvilke kolonnekategorier vi har
pingvindata.columns
# Teller ulike forekomster i kolonnen
pingvindata["sex"].value_counts()
# Finne en spesifikk kolonne
pingvindata["flipper_length_mm"]
# Finne et utvalg elementer [fra:til] av en spesifikk kolonne
pingvindata["flipper_length_mm"][0:10]
# Finne flere kolonner
pingvindata[["bill_length_mm", "bill_depth_mm", "flipper_length_mm"]]
# Finne spesifikke elementer
pingvindata.loc[1] # Element 1 (andre element)
pingvindata.loc[100][2] # Element 100, kolonneverdi 2
pingvindata[pingvindata["sex"] == "FEMALE"] # Damepingviner
# Sortere verdier etter stigende (ascending = True) rekkefølge (først nebblengde, så nebbdybde)
pingvindata.sort_values(["bill_length_mm", "bill_depth_mm"], ascending = True)
# Velger ut kun de pingvinene med nebblengde under 40 mm
pingvindata_kort_nebb = pingvindata[pingvindata["bill_length_mm"] < 40]
# Sletter alle pingviner som har mangelfull info (NaN)
pingvindata.dropna()
# Legger til en ny kolonne
pingvindata["total_mm"] = pingvindata["bill_length_mm"] + pingvindata["bill_depth_mm"]
# Lagre fila i en ny csv-fil
pingvindata.to_csv("ny_pingvinfil.csv", index = False)
# Sletter kolonnen har lagt til
pingvindata.pop("total_mm")
pingvindata.head()
| species | island | bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | sex | |
|---|---|---|---|---|---|---|---|
| 0 | Adelie | Torgersen | 39.1 | 18.7 | 181.0 | 3750.0 | MALE |
| 1 | Adelie | Torgersen | 39.5 | 17.4 | 186.0 | 3800.0 | FEMALE |
| 2 | Adelie | Torgersen | 40.3 | 18.0 | 195.0 | 3250.0 | FEMALE |
| 3 | Adelie | Torgersen | NaN | NaN | NaN | NaN | NaN |
| 4 | Adelie | Torgersen | 36.7 | 19.3 | 193.0 | 3450.0 | FEMALE |
Underveisoppgave
Bruk pingvindatafila (du finner den under datafiler på nettsidene her) og les dataene med Pandas. Finn ut hvor mange hankjønnspingviner det er med kroppsmasse under 3000 g.
Løsningsforslag
En mulig løsning er å gjøre det slik, eller kun sortere kroppsmasse og så bruke count_values til å telle antall av hvert kjønn.
data1 = pingvindata[pingvindata["sex"] == "MALE"]
data1 = data1[data1["body_mass_g"] < 3500]
print("Antall hankjønnspinginver:", len(data1))
Underveisoppgave
Vi kan beskrive frekvens som antall forekomster av en verdi som tilhører en variabel. Relativ frekvens er antallet forekomster av noe delt på totalt antall verdier innenfor den samme variabelen. Regn ut relativ frekvens av antall pingviner av arten Adelie.
Løsningsforslag
En mulig løsning er å gjøre det slik, eller kun sortere kroppsmasse og så bruke count_values til å telle antall av hvert kjønn.
arter = pingvindata["species"].value_counts()
print(arter) # ser at Adelie er element 0 i lista over antall arter
adelie = arter[0]
n = len(pingvindata)
print("Relativ frekvens:", round(adelie/n,2))
Visualisering#
Visualisering av data er viktig. Det gir oss mulighet til å enkelt se sammenhenger og tolke data. En illustrasjon bør være så klar og enkel som mulig, uten unødvendige detaljer. Vær derfor kritisk når du skal lage dine egne figurer. Vi skal bruke et bibliotek som heter seaborn til å lage fine visualiseringer. Seaborn-biblioteket inneholder noen ekstra fine plott som utvidelse til matplotlib, og vi kan bruke vanlige matplotlib-kommandoer som xlabel og title sammen med seaborn-kommandoene. La oss på noen få nyttige plott her. Vi skal se nærmere på andre plott ettersom de blir nyttige.
Her er et eksempel på et relasjonsplott (“relplot”), som viser sammenhengen mellom to variabler. Vi kan lage ulike markører og modifisere størrelsen til markørene. I plottet nedenfor er det kanskje litt i overkant mye informasjon som formidles, men det viser i hvert fall hva som er mulig.
import matplotlib.pyplot as plt
import seaborn as sns
sns.relplot(data=pingvindata, x="bill_length_mm",y="bill_depth_mm", hue="species", size="body_mass_g", style="island")
plt.xlabel("Nebblengde (mm)")
plt.ylabel("Nebbdybde (mm)")
plt.title("Sammenheng mellom nebbdybde og nebblengde hos pingviner")
plt.show()
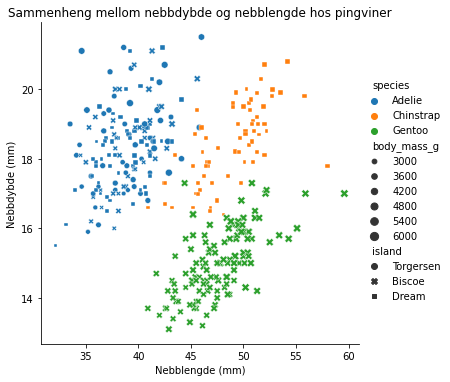
Underveisoppgave
Forklar hva plottet ovenfor formidler. Foreslå eventuelt forbedringer av visualiseringen.
Visualiseringen nedenfor formidler noe av det samme, men med fokus på hvilke verdier som er mest vanlig hos de ulike artene. Vi kaller det for et “tetthetsplott” (kde = kernel density estimate), siden det viser fordelingen (tettheten) av de ulike verdiene. En slik visualisering kan leses omtrent som et kart med høydenivåer - den innerste “ringen” viser området der det er “høyest”, altså har flesteparten av pingvinene denne fordelingen.
sns.jointplot(data=pingvindata, x="bill_length_mm",y="bill_depth_mm", hue="species", kind = "kde")
<seaborn.axisgrid.JointGrid at 0x2c48001ea60>
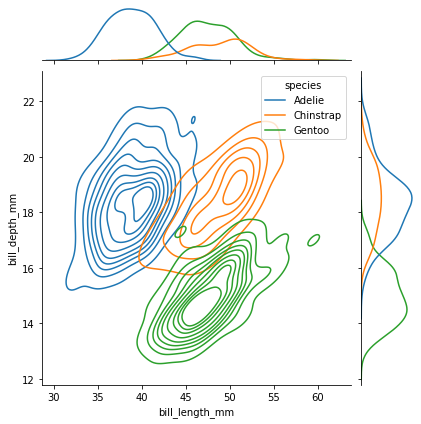
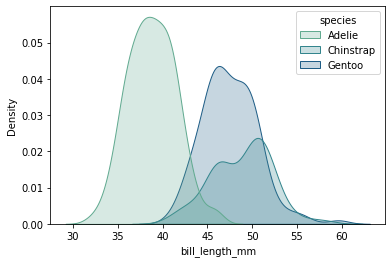
Plottet nedenfor er en variant av et tetthetsplott for én variabel:
sns.kdeplot(data=pingvindata, x="bill_length_mm", hue="species", fill=True, palette="crest")
<AxesSubplot:xlabel='bill_length_mm', ylabel='Density'>
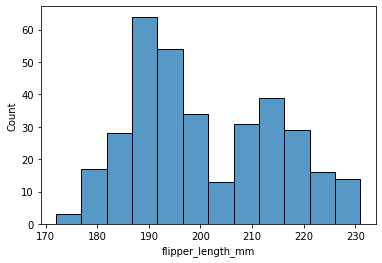
En annen nyttig visualisering er histogrammer. Det er figurer som viser fordeling av verdier ved å gruppere ulike verdier i intervaller . Du kan velge hvor mange søyler/intervaller som skal være med i histogrammet ved å justere parameteren bins.
sns.histplot(data=pingvindata, x='flipper_length_mm', bins = 12)
<AxesSubplot:xlabel='flipper_length_mm', ylabel='Count'>
Som sagt finnes det mange andre mulige visualiseringer og måter å pynte plottene på, men vi nøyer oss i første omgang med disse.
Oppgaver#
Oppgave
Den molare massen til et grunnstoff kan beregnes ved å ta summen av den molare massen til hvert av isotopene til grunnstoffet multiplisert med andelen/forekomsten av denne istopen:
Her er \(m_i\) massen til den \(i\)-te isotopen av grunnstoffet, og \(w_i\) er forekomsten av denne istopen (fra 0 til 1, der 1 er 100 prosent forekomst). Fila tinn.txt inneholder forekomsten i % av ulike isotoper av tinn, som er grunnstoffet med mest stabile isotoper.
Les av fila og legg nuklidetallet i en liste, massen i en annen liste og forekomsten i prosent/100 i en tredje liste.
Bruk informasjonen til å regne ut den molare massen til tinn. Print ut massen med fire desimaler og korrekt enhet.
Legg inn en feilhåndtering som gir en feil dersom fila ikke kan leses.
Oppgave
Fila titrering.txt inneholder en kolonne med tilsatt volum NaOH i mL til en løsning med eddiksyre og en kolonne med pH.
Les av fila og lag to lister eller arrayer med navn volum og pH med de aktuelle verdiene i. Plott titrerkurven.
I videoene nedenfor kan du få en innføring eller repetisjon i den grunnleggende teorien bak lesing av filer:
